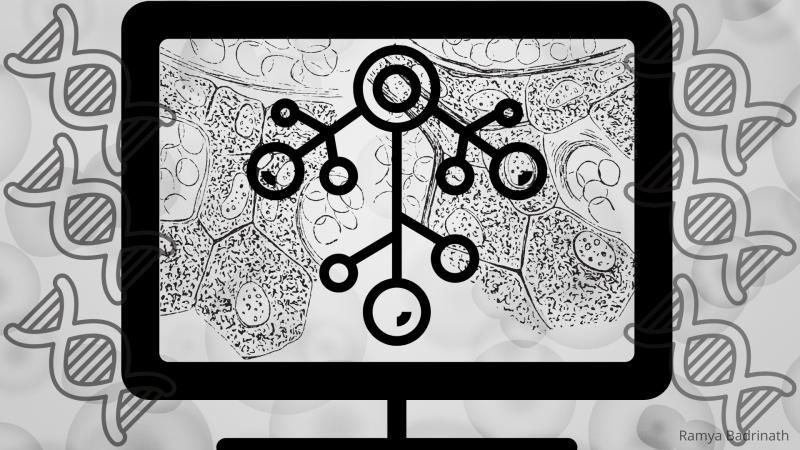
नेचर कम्युनिकेशंस में प्रकाशित एक हालिया अध्ययन में, शोधकर्ताओं ने कोशिकीय वंशावली के पुनर्निर्माण के लिए एक नई सांख्यिकीय विधि, ‘LinTIMaT’ (लिनटीआईएमटी) का वर्णन किया है, जो वैज्ञानिकों को परस्पर विकसित होती हुई जैविक प्रणाली में कोशिकाओं के विकास को समझने की क्षमता प्रदान करती है।
कोशिकाएँ हमारे जैसी हैं; वे पूर्णतया कार्यशील इकाइयाँ हैं जो अपने पूर्वजों से विकसित हुई हैं, और इसलिए, हमारी तरह, उनके पास भी एक पारिवारिक पेड़ अर्थात् एक जाति वंशवृक्ष है। जीवों के विकास, विभिन्न रोग, एवं जीव विज्ञान में आने वाले कई सवालों के जवाब खोजने के लिए वैज्ञानिक इन जाति वंशवृक्षों का उपयोग कर कोशिकाओं और उनके पूर्वजों के बीच संबंधों का पता लगाते हैं।
जाति वंशवृक्षों के निर्माण के लिए कोशिकीय वंशावली का ज्ञान होना अत्यावश्यक है - कौन सी कोशिका किस से और कैसे विकसित हुई! कोशिका वंशों का वर्णन करना चुनौतीपूर्ण है क्योंकि सबसे सरल जीवों में भी हजारों कोशिकाएँ होती हैं। अतः किसी भी जीव की हर एक कोशिका अपनी पैतृक कोशिका से कैसे विकसित होती है, इसका पता लगाने के लिए बहुत सी जानकारी की आवश्यकता होती है। इसी समस्या का समाधान करने में उच्च-थ्रूपुट क्षमता वाली अनुक्रमण तकनीक और सांख्यिकीय विश्लेषण काम आता है।
LinTIMaT अनेकों प्रकार के उत्परिवर्तन एवं आनुवांशिक अभिव्यक्ति डेटा को एकीकृत कर डेटा और यंत्र अधिगम के माध्यम से कोशिकीय वंशावली का पुनर्निर्माण करने में सक्षम है। यह एल्गोरिदम उन मौजूदा तरीकों की तुलना में बेहतर एवं सटीक परिणाम देता है, जो आनुवांशिक उत्परिवर्तन या अभिव्यक्ति डेटा का अलग-अलग प्रयोग करते हैं।
अपने व्यावहारिक प्रयोग के कारण LinTIMaT की संभावना-आधारित पद्धति का निस्संदेह दुनिया भर के जीवविज्ञानी प्रयोग करना चाहेंगे। इस पद्धति का उपयोग कई अनुसंधानों में किया जा सकता है जिनमें से एक है, कैंसर। कैंसर के ऊतक विविध प्रकार के बने होते हैं। संभवत यह सभी ऊतक एक ही कोशिका से उत्पन्न होते हैं। कैंसर ऊतकों के कोशिकीय वंशवृक्षों के निर्माण से चिकित्सकों को इस बीमारी के उपचार के लिए एक ऐसी सूचित चिकित्सा प्रणाली बनाने में सहायता मिलेगी जिसमें उन दवाओं का प्रयोग हो जो कोशिकाओं की एक विशेष श्रेणी पर काम करें।
सितंबर 2018 में डॉ. हमीद ज़फर, जो अब भारतीय प्रोध्योगिक संस्थान कानपुर में एक सहायक प्राध्यापक हैं, पोस्टडॉक्टोरल फेलो के रूप में कार्नेगी मेलन विश्वविद्यालय में काम किया था। वहाँ डॉ. ज़फ़र ने कंप्यूटर विज्ञान एवं एकल कोशिकाओं से डीएनए अनुक्रमण डेटा का उपयोग करते हुए जाति वंशवृक्ष निर्माण में विशेषज्ञता प्राप्त की। अनुक्रमण कुछ रसायनों और कंप्यूटर एल्गोरिदम का उपयोग कर डीएनए अथवा आरएनए में न्यूक्लियोटाइड के क्रम को उजागर करता है।
कार्नेगी मेलन में अपने शोध के दौरान, डॉ. ज़फ़र ने महसूस किया कि एकल-कोशिका आरएनए (scRNA) अनुक्रमण तकनीक अब जीव-विज्ञान और चिकित्सा क्षेत्र में अपने अनुप्रयोगिक मूल्यों के लिए लोकप्रियता प्राप्त कर रही थी। अतः वह अपनी विशेषज्ञता का प्रयोग कर सिंगल-सेल आरएनए सीक्वेंसिंग डेटा पर काम करना चाहते थे, इसी दौरान उन्होंने देखा की जाति वंशवृक्ष पुनर्निर्माण के मौजूदा तरीकों में कुछ कमियाँ थीं। उन्होंने और उनके पोस्टडॉक्टोरल मेंटर प्राध्यापक ज़ीव बार-जोसेफ ने यह पता लगाया कि उत्परिवर्तन और अभिव्यक्ति डेटासेट का संयोजन इसका उत्तर हो सकता है। डॉ. ज़फ़र ने प्राध्यापक बार-जोसेफ की प्रयोगशाला के एक छात्र चीह लिन के साथ मिलकर LinTIMaT विकसित किया और इसकी क्षमता का अन्वेषण किया। कार्नेगी मेलन में अपने पोस्टडॉक्टोरल कार्यकाल के बाद, डॉ. ज़फ़र ने इस परियोजना को समुद्र के उस पार भारतीय प्रौद्योगिकी संस्थान कानपुर तक पहुँचाया, जहाँ इस कार्य को पूरा किया गया।
LinTIMaT दो प्रकार के डेटासेट का उपयोग करता है। पहला एक कोशिका उत्परिवर्तन डेटा सेट है, जो उन कोशिकाओं से प्राप्त डेटा के साथ काम करता है जिनमें मार्कर सरणी समाविष्ट हैं। जैसे-जैसे कोशिकाएँ विभाजित और विकसित होती हैं, ये सरणियाँ उत्परिवर्तन जमा करती हैं, जो इस संदर्भ में CRISPR-Cas9 नामक एक लोकप्रिय आनुवंशिक इंजीनियरिंग प्रणाली द्वारा समाविष्ट की जाती हैं। उत्परिवर्तन डेटा, जिसमें यह जानकारी हो की किन कोशिकाओं में किस प्रकार का उत्परिवर्तन हुआ एवं कब, हमें बहुत कुछ बता सकता है। वैज्ञानिक हजारों कोशिकाओं से यह जानकारी एकत्र करते हैं और उन्हें कंप्यूटर एल्गोरिदम के माध्यम से समझते हैं।
LinTIMaT में उपयोग होने वाला दूसरे प्रकार के डेटा जीन अभिव्यक्ति मूल्य हैं । यह scRNA अनुक्रमण डेटा है जो व्यक्तिगत कोशिकाओं के आरएनए अभिव्यक्ति प्रोफाइल प्रदान करता है। डेटा, जो उच्च-थ्रूपुट है, कोशिकाओं के प्रकार को समझने में सहायता करता है और इस प्रकार कोशिका वंश के बिना जोड़ी के टुकड़ों की पहेली को बेहतर तरीके से जोड़ने में मदद करता है। इस डेटा का प्रयोग केवल कोशिका उत्परिवर्तन की जानकारी के आधार पर किए जाने वाले वंशवृक्ष पुनर्निर्माण की अस्पष्टता को कम करने में मदद करता है।
प्रायोगिक जीवविज्ञानी मैक्सिमम पारसीमोनी नामक एक शास्त्रीय, जाति वंशवृक्ष पुनर्निर्माण विधि का उपयोग करते हैं, जो हालाँकि अत्यधिक मूल्यवान है, लेकिन इसमें कुछ कमियाँ हैं। डॉ. ज़फ़र बताते हैं कि, “यह केवल आनुवंशिक मार्करों के आधार पर वंश को फिर से संगठित करता है। इस तरह के दृष्टिकोण से, हम कुछ कोशिका वंश की कुछ शाखाओं को पुनर्प्राप्त करने में सक्षम नहीं होंगे, क्योंकि कोई भी आनुवंशिक मार्कर उन में उत्परिवर्तन के साथ समर्थित नहीं हैं और इसलिए हमें अधूरी जानकारी प्राप्त होती है। एक और दोष यह है कि हम आनुवंशिक मार्कर का उपयोग करने वाले व्यक्तियों के डेटा को एकीकृत नहीं कर सकते हैं, जो पूरी तरह से यादृच्छिक हैं।"
वर्तमान में scRNA अनुक्रमण डेटा का उपयोग कोशिकाओं के विभेदक प्रक्षेपवक्र को संदर्भित करने के लिए किया जा रहा है। फिर भी समस्या यह है कि आपको जो मिलता है वह सटीक आनुवांशिक वंशावली नहीं है बल्कि एक ऐसा निरूपण है जो दर्शाता है कि कैसे जीन अभिव्यक्ति का मान एक कोशिकीय अवस्था से दूसरे में कैसे परिवर्तित होता है। हालाँकि इन विधियों में कई खूबियाँ हैं, लेकिन इनकी सीमाओं और कमज़ोरियों को अनदेखा नहीं किया जा सकता।
डॉ. ज़फ़र का मानना है की, "LinTIMaT के साथ, हम इन कमियों को दरकिनार कर सकते हैं। वर्तमान में उपयोग किए जाने वाले तरीकों की तुलना में, यह एक ऐसा एकल कंप्यूटर आधारित फ्रेमवर्क जिससे कोशिका जाति वंशवृक्ष पुनर्निर्माण को अधिक सटीक रूप से किया जा सकता है।" सिंगल-सेल ट्रांसक्रिपटोमिक डेटा को एकीकृत करके, LinTIMaT केवल उत्परिवर्तन डेटा से प्राप्त सेल वंश परिदृश्य में खामियों को दूर करता है।
"यह देखना सबसे रोमांचक था कि इन दो अलग-अलग डेटा सेटों का एक साथ उपयोग कर, जो कि पहले किसी ने नहीं किया था, हमें वास्तव में मौजूदा तरीकों की तुलना में कई गुना बेहतर परिणाम दे रहा था।” इसके अलावा, LinTIMaT एक सुसंगत वंश वृक्ष के पुनर्निर्माण के लिए कई अलग-अलग वंशों के एकीकरण की अनुमति देता है।
अंत में अपने बनाए इस मॉडल को अनुकूलित कर आगे बढ़ने के संकेत देते हुए डॉ. ज़फ़र कहते हैं, "मैं अब और अधिक व्यापक तरीके से इस मॉडल पर काम करना चाहता हूँ जो हमारे परिणामों में और भी सुधार करेगा।”